产品概述
—
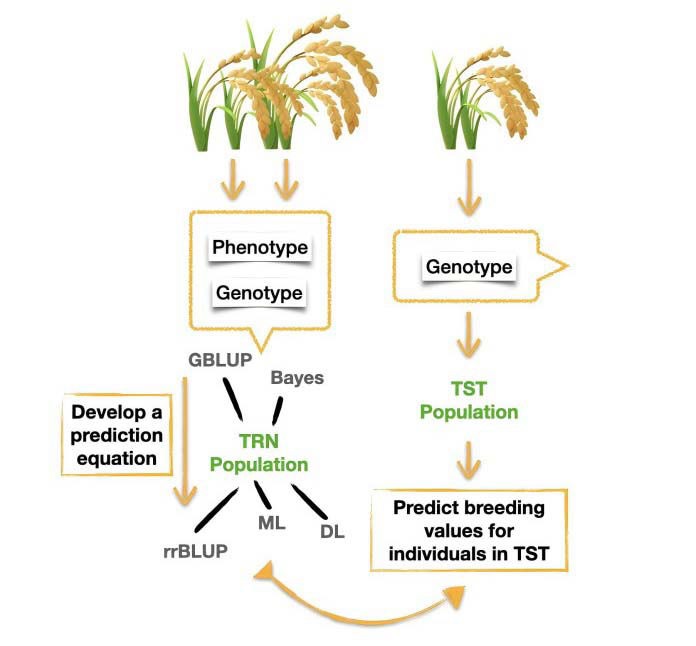
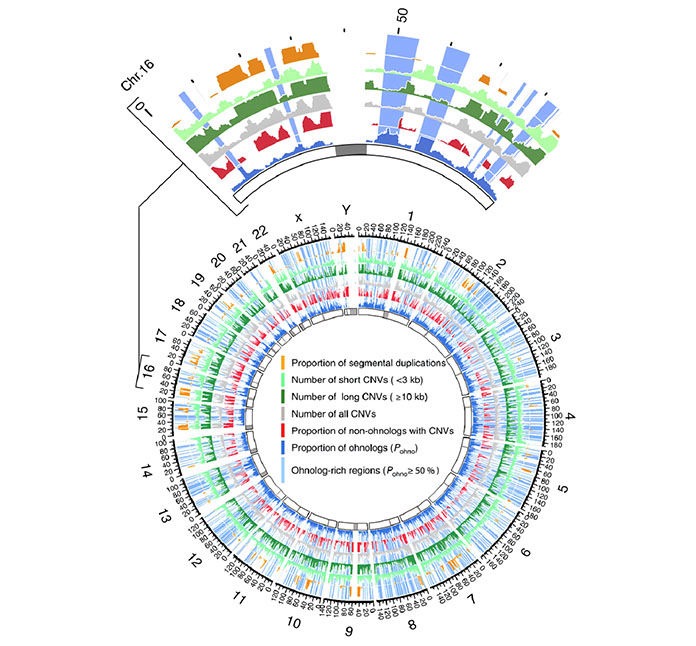
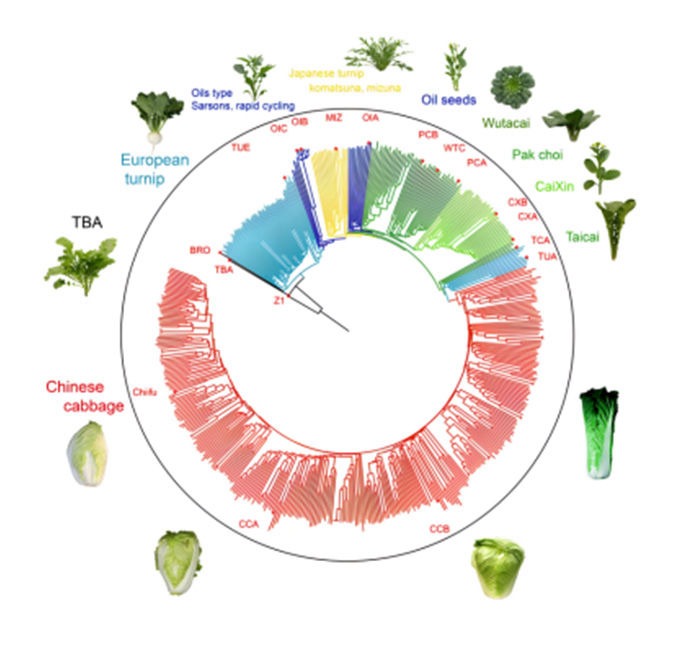
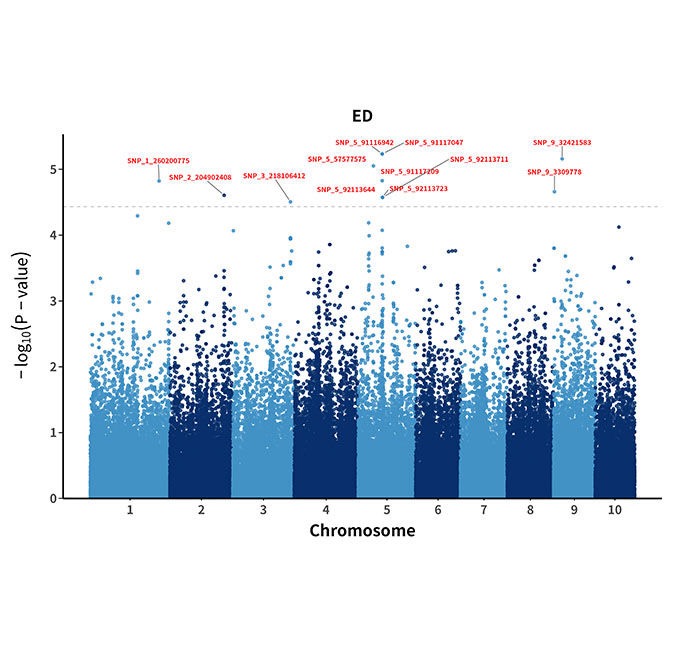
GSBrain是一款基于模块化和通用性设计为原则的可视化多组学AI分析平台,用户通过简单拖拽连接即可实现基因组等数据的分析评估,使得用户即使是非计算机背景也可轻松对多组学数据运用深度学习等技术进行处理和挖掘,功能包括全基因组选择(GS)、全基因组关联分析(GWAS)、基因同源度分析(HA)、拷贝数变异分析(CNV)、基因存在缺失变异分析(PAV)、启动子顺式调控元件(CRE)以及任意组学的DL分析挖掘等。此外,平台支持私有化部署,以保障用户数据隐私安全,并可通过云端同时支持多用户。